רצף חוזר
רצפים חוזרים (הידועים גם כאלמנטים חוזרים, יחידות חוזרות או חזרות) הם תבניות קצרות או ארוכות של חומצות גרעין (DNA או RNA) המופיעות במספר עותקים ברחבי הגנום. באורגניזמים רבים, חלק ניכר מה-DNA חוזר על עצמו, כאשר למעלה משני שלישים מהרצף מורכב מאלמנטים חוזרניים בבני אדם.[1] חלק מהרצפים החוזרים הללו נחוצים לשמירה על מבני גנום חשובים כגון טלומרים.
רצפים חוזרים מסווגים לסיווגים שונים לפי תכונות כגון מבנה, אורך, מיקום, מוצא ואופן ההכפלה.[2]
בעוד שרצפי DNA חוזרים מסוימים חשובים לתפקוד הסלולרי ולתחזוקת הגנום, רצפים חוזרים יכולים גם לפגוע. רצפי DNA רבים שחוזרים על עצמם נקשרו למחלות אנושיות כמו מחלת הנטינגטון ואטקסיה של פרידרייך. חלק מהאלמנטים החוזרים על עצמם הם נייטרליים ומתרחשים כאשר אין העדפה עבור רצפים ספציפיים בהתאם לאופן שבו מתרחשת טרנספוזיציה או מעבר.[3] עם זאת, שפע של חזרות נייטרליות יכול להשפיע על התפתחות הגנום כפי שהם מצטברים עם הזמן. בסך הכל, רצפים חוזרים הם תחום מיקוד חשוב מכיוון שהם יכולים לספק תובנה לגבי מחלות אנושיות והתפתחות הגנום.[3]
היסטוריה של גילוי
בשנות ה-50, ברברה מקלינטוק צפתה לראשונה בשינוי מיקום של DNA והמחישה את תפקידי הצנטרומר והטלומר בסימפוזיון Cold Spring Harbor.[4] עבודתו של מקלינטוק הכינה את הבמה לגילוי רצפים חוזרים, מכיוון שהטרנספוזיציה, מבנה הצנטרומר ומבנה הטלומרים נוצרו כולם באמצעות אלמנטים שחוזרים על עצמם, אך הדבר לא הובן במלואו באותה תקופה. המונח "רצף חוזר" שימש לראשונה על ידי רוי ג'ון בריטן ו-DE Kohne ב-1968; הם גילו שיותר ממחצית מהגנומים האאוקריוטים (עם גרעין) היו עם DNA שחוזר על עצמו באמצעות הניסויים שלהם על חיבור מחדש של DNA.[5] למרות שרצפי ה-DNA החוזרים ונשנים נשמרו ונמצאו בכל מקום, תפקידם הביולוגי עדיין לא היה ידוע. בשנות ה-90, מחקר נוסף נערך כדי להבהיר את הדינמיקה האבולוציונית של חזרות minisatellite וmicrosatellite בגלל חשיבותם בזיהוי פלילי מבוסס-DNA ואקולוגיה מולקולרית. חזרות מפוזרות ב-DNA הוכרו יותר ויותר כמקור פוטנציאלי לשונות ורגולציה גנטית. גילויים של מחלות חוזרות ונשנות הקשורות ל-DNA חוזר עוררו עניין נוסף בתחום המחקר הזה.[6] בשנות ה-2000, הנתונים מרצף הגנום האאוקריוטי המלא (פרויקט הגנום האנושי) אפשרו זיהוי של מקדמים, משפרים ו-RNA רגולטוריים שונים אשר כולם מקודדים על ידי אזורים חוזרים. כיום, התפקידים המבניים והרגולטוריים של רצפי DNA חוזרים ונשנים נותרו תחום מחקר פעיל.
סוגים ופונקציות
רצפים חוזרים רבים צפויים להיות שרידים לא פונקציונליים, שאריות של אלמנטים ניתנים להעברה, אלה סומנו כ"זבל" או "אנוכי" DNA.[7][8][9] עם זאת, לפעמים כמה חזרות עשויות להיות הרחבה של פונקציות אחרות.[10]
חזרות טנדם הן רצפים חוזרים ונשנים שצמודים זה לזה ישירות בגנום.[11] חזרות טנדם עשויות להשתנות במספר הנוקלאוטידים המרכיבים את הרצף החוזר, כמו גם במספר הפעמים שהרצף חוזר על עצמו. כאשר הרצף החוזר הוא באורך של 2–10 נוקלאוטידים בלבד, החזרה מכונה חזרת טנדם קצרה (STR) או microsatellite..[12] כאשר הרצף החוזר הוא באורך 10–60 נוקלאוטידים, החזרה מכונה minisatellite.[13] עבור minisatellite וmicrosatellite, מספר הפעמים שהרצף חוזר בלוקוס בודד יכול לנוע בין פעמיים למאות פעמים.
לחזרות טנדם יש מגוון רחב של פונקציות ביולוגיות בגנום. לדוגמה, מיני לוויינים הם לעיתים קרובות נקודות חמות של רקומבינציה הומולוגית מיוטית באורגניזמים אוקריוטיים.[14] רקומבינציה היא כאשר שני כרומוזומים הומולוגיים מתיישרים, נשברים ומתחברים מחדש כדי להחליף חלקים. הרקומבינציה חשובה כמקור לגיוון גנטי, כמנגנון לתיקון DNA פגום וכצעד הכרחי בהפרדה מתאימה של כרומוזומים במיוזה.[14] הנוכחות של DNA ברצף חוזר מקלה על אזורי הומולוגיה להתיישר, ובכך לשלוט מתי והיכן מתרחש רקומבינציה.
בנוסף לתפקיד החשוב ברקומבינציה, חזרות טנדם ממלאות גם תפקידים מבניים חשובים בגנום. לדוגמה, הטלומרים מורכבים בעיקר מחזרות טנדם של TTAGGG.[15] חזרות אלו מתקפלות למבני G quadruplex מאורגנים מאוד אשר מגנים על קצוות ה-DNA הכרומוזומלי מפני השפלה.[16] אלמנטים חוזרים מועשרים גם באמצע הכרומוזומים. צנטרומרים הם האזורים הדחוסים ביותר של כרומוזומים המחברים את הכרומטידות האחיות יחדיו ומאפשרים גם לציר המיטוטי להיצמד ולהפריד בין כרומטידות אחיות במהלך חלוקת התא.[17] צנטרומרים מורכבים מחזרת טנדם של 177 זוגות בסיסים בשם החזרה של α-לווין.[16] הטרוכרומטין פריצנטרומרי, ה-DNA המקיף את הצנטרומר וחשוב לתחזוקה מבנית, מורכב מתערובת של תת-משפחות לוויינים שונות כולל הלוויינים α-, β- ו-γ וכן חזרות של HSATII, HSATIII ו-sn5.[18][19]

חלק מהרצפים שחוזרים על עצמם, כמו אלה בעלי תפקידים מבניים שנדונו לעיל, ממלאים תפקידים הנחוצים לתפקוד ביולוגי תקין. לחזרות טנדם אחרות יש תפקידים מזיקים הגורמים מחלות. עם זאת, לחזרות טנדם רבות אחרות יש פונקציות לא ידועות או לא מובנות.[20]
חזרות משובשות
חזרות משובשות הן רצפי DNA זהים או דומים שנמצאים במקומות שונים ברחבי הגנום.[21] חזרות רצופות מובדלות מחזרות טנדם בכך שהרצפים החוזרים אינם סמוכים זה לזה ישירות אלא עשויים להיות מפוזרים בין כרומוזומים שונים או רחוקים זה מזה על אותו כרומוזום. רוב החזרות המשולבות הן אלמנטים ניתנים להפיכה (TEs), רצפים ניידים שניתן "לחתוך ולהדביק" במקומות שונים בגנום.[22] TEs נקראו במקור "גנים קופצים" בשל יכולתם לנוע, אולם המונח הזה מטעה במידה מסוימת מכיוון שלא כל ה-TEs הם גנים יחידים.[23]
אלמנטים הניתנים להעברה המתועתקים ל-RNA, מתעתקים לאחור ל-DNA, ואז משתלבים מחדש בגנום נקראים רטרוטרנספוזונים.[22] בדיוק כפי שחזרות טנדם מסווגות עוד יותר על סמך אורך הרצף החוזר, ישנם סוגים רבים ושונים של רטרוטרנספוזונים. אלמנטים גרעיניים ארוכים ( LINEs ) הם בדרך כלל באורך של 3–7 קילו-בסיסים.[24] אלמנטים גרעיניים קצרים ( SINEs ) הם בדרך כלל 100–300 זוגות בסיסים ולא יותר מ-600 זוגות בסיסים.[24] רטרוטרנספוזונים חוזרים ארוכי קצה (LTR) הם מחלקה עיקרית שלישית של רטרוטרנספוזונים ומאופיינים ברצפים שחוזרים על עצמם מאוד כקצוות החזרה.[22] כאשר יסוד שניתן להעברה אינו עובר דרך RNA כחומר ביניים, זה נקרא טרנספוזון DNA.[22]
על פי הערכות, יסודות הניתנים להעברה מהווים 45% מהגנום האנושי.[25] מאז התפשטות בלתי מבוקרת של TEs עלולה להמיט הרס על הגנום, מנגנוני רגולציה רבים התפתחו כדי להשתיק את התפשטותם, כולל מתילציה של DNA, שינויים היסטונים, RNAs לא מקודדים (ncRNAs) כולל RNA מתערב קטן (siRNA), משפצי כרומטין, גרסאות היסטונים, וגורמים אפיגנטיים אחרים.[23] עם זאת, TEs ממלאים מגוון רחב של פונקציות ביולוגיות חשובות. כאשר TEs מוכנסים למארח חדש, כגון מנגיף, הם מגבירים את המגוון הגנטי.[23] במקרים מסוימים, אורגניזמים מארחים מוצאים פונקציות חדשות עבור החלבונים הנובעות מביטוי TEs בתהליך אבולוציוני הנקרא TE exaptation.[23] מחקרים אחרונים מראים גם ש-TEs משמשים לשמירה על מבנה כרומטין מסדר גבוה וארגון הגנום התלת-ממדי.[26] יתר על כן, TEs תורמים לוויסות הביטוי של גנים אחרים בכך שהם משמשים כמשפרים דיסטליים ואתרי קשירה של גורמי שעתוק.[27]
השכיחות של אלמנטים משולבים בגנום משכה תשומת לב למחקר נוסף על מקורותיהם ותפקידיהם. אופיינו כמה אלמנטים ספציפיים המשולבים, כמו החזרה של Alu ו-LINE1.
חזרות ישירות והפוכות
בעוד שחזרות טנדם והפוכות מובדלות על סמך מיקומן בגנום, חזרות ישירות והפוכות מובדלות על סמך הסדר של בסיסי הנוקלאוטידים. חזרות ישירות מתרחשות כאשר רצף נוקלאוטידים חוזר על עצמו באותה כיווניות. חזרות הפוכות מתרחשות כאשר רצף נוקלאוטידים חוזר על עצמו בכיוון ההפוך. לדוגמה, חזרה ישירה של "CATCAT" תהיה חזרה נוספת של "CATCAT". לעומת זאת, החזרה ההפוכה תהיה "TACTAC". כאשר אין נוקלאוטידים המפרידים בין החזרה ההפוכה, כגון "CATCATTACTAC", הרצף נקרא חזרה פלינדרום. חזרות הפוכות יכולות למלא תפקידים מבניים ב-DNA וב-RNA על ידי יצירת לולאות גזע וצלבים.[28]
רצפים חוזרים במחלות אנושיות
עבור בני אדם, כמה רצפי DNA חוזרים ונשנים קשורים למחלות. באופן ספציפי, רצפים חוזרים בטנדם, עומדים בבסיס מספר מצבי מחלה אנושיים, במיוחד מחלות חוזרות של טרינוקלאוטידים כמו מחלת הנטינגטון, תסמונת X שביר, מספר אטקסיות ספינו - מוחיות, ניוון מיוטוני ואטקסיה של פרידרייך.[29] התרחבות חוזרת של טרינוקלאוטידים בקו הנבט לאורך דורות עוקבים עלולה להוביל לביטויים חמורים יותר ויותר של המחלה. הרחבות חוזרות אלו של טרינוקלאוטיד עשויות להתרחש באמצעות החלקת גדיל במהלך שכפול DNA או במהלך סינתזת תיקון DNA.[29] צוין כי גנים המכילים CAG חוזרים פתוגניים מקודדים לרוב לחלבונים שיש להם בעצמם תפקיד בתגובת נזק ל-DNA וכי הרחבות חוזרות עלולות לפגוע במסלולי תיקון DNA ספציפיים.[30] תיקון שגוי של נזקי DNA ברצפים חוזרים עלול לגרום להרחבה נוספת של רצפים אלו, ובכך ליצור מעגל קסמים של פתולוגיה.[30]
מחלת הנטינגטון
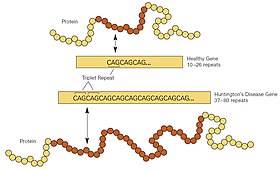
מחלת הנטינגטון היא הפרעה נוירודגנרטיבית הנובעת מהתרחבות של CAG ברצף טרינוקלאוטידים חוזר באקסון 1 של הגן האנטינגטין ( HTT ). גן זה אחראי על קידוד החלבון huntingtin אשר ממלא תפקיד במניעת אפופטוזיס,[31] הידוע גם בשם מוות תאי, ותיקון נזק ל-DNA חמצוני.[32] במחלת הנטינגטון ההרחבה של רצף הטרינוקלאוטידים CAG מקודדת לחלבון האנטינגטין מוטנטי עם תחום פוליגלוטמין מורחב.[33] תחום זה גורם לחלבון ליצור אגרגטים בתאי עצב המונעים תפקוד תאי תקין וכתוצאה מכך לניוון עצבי.

תסמונת ה-X השביר
תסמונת X שביר נגרמת מהרחבת רצף ה-DNA CCG בגן FMR1 על כרומוזום X.[34] גן זה מייצר את החלבון קושר RNA FMRP. במקרה של תסמונת X שביר הרצף החוזר הופך את הגן לבלתי יציב ולכן משתיק את הגן FMR1.[35] מכיוון שהגן שוכן על כרומוזום X, נשים שיש להן שני כרומוזומי X מושפעות פחות מזכרים שיש להם רק כרומוזום X וכרומוזום Y אחד, מכיוון שכרומוזום X השני יכול לפצות על השתקת הגן בכרומוזום X השני.
אטקסיה של Spinocerebellar
למחלה spinocerebellar ataxias יש רצפים חוזרים של טרינוקלאוטידים של CAG העומדים בבסיס מספר סוגים של אטקסיות spinocerebellar (SCAs - SCA1 ; SCA2; SCA3; SCA6; SCA7; SCA12; SCA17 ).[36] בדומה למחלת הנטינגטון, זנב הפוליגלוטמין שנוצר עקב התרחבות טרינוקלאוטיד זו גורם לצבירה של חלבונים, מונע תפקוד תאי תקין וגורם לניוון עצבי.[37]
האטקסיה של פרידרייך
אטקסיה של פרידרייך היא סוג של אטקסיה שיש לה רצף חוזר מורחב GAA בגן frataxin.[38] הגן frataxin אחראי לייצור חלבון frataxin, שהוא חלבון מיטוכונדריאלי המעורב בייצור אנרגיה ובנשימה התאית.[39] רצף ה-GAA המורחב מביא להשתקת האינטרון הראשון וכתוצאה מכך לאובדן תפקוד בחלבון הפרטקסין. אובדן של גן FXN תפקודי מוביל לבעיות בתפקוד המיטוכונדריאלי בכללותו ויכול להופיע באופן פנוטיפי בחולים כקושי בהליכה.
ניוון מיוטוני
ניוון מיוטוני היא הפרעה המתבטאת כחולשת שרירים ומורכבת משני סוגים עיקריים: DM1 ו-DM2.[40] שני סוגי ניוון מיוטוניים נובעים מרצפי DNA מורחבים. ב-DM1 רצף ה-DNA שמתרחב הוא CCG ואילו ב-DM2 הוא CCTG. שני רצפים אלו נמצאים על גנים שונים כאשר הרצף המורחב ב-DM2 נמצא בגן ZNF9 והרצף המורחב ב-DM1 נמצא בגן DMPK. שני הגנים אינם מקודדים לחלבונים בניגוד להפרעות אחרות כמו מחלת הנטינגטון או תסמונת ה-X השביר. עם זאת, הוכח שיש קשר בין רעילות RNA לבין הרצפים החוזרים ב-DM1 ו-DM2.
טרשת צדדית אמיוטרופית ודמנציה פרונטוטמפורלית
לא כל המחלות הנגרמות על ידי רצפי DNA חוזרים הם מחלות חוזרות של טרינוקלאוטידים. המחלות amyotrophic lateral sclerosis ו-frontotemporal דמנציה נגרמות על ידי רצפים חוזרים של hexanucleotide GGGGCC בגן C9orf72, הגורמים לרעילות RNA המובילה לניוון עצבי.[41][36]
ביוטכנולוגיה
קשה לרצף DNA חוזר באמצעות טכניקות רצף מהדור הבא מכיוון שהרכבת רצף מקריאות קצרות פשוט לא יכולה לקבוע את אורכו של חלק שחוזר על עצמו. בעיה זו חמורה במיוחד עבור מיקרו-לוויינים, העשויים מיחידות חוזרות זעירות של 1-6bp.[42] למרות שקשה לרצף אותם, לחזרות הקצרות הללו יש ערך רב בטביעת אצבע של DNA ובמחקרים אבולוציוניים. חוקרים רבים השאירו היסטורית רצפים חוזרים בניתוח ופרסום נתוני גנום שלם בשל מגבלות טכניות.[43]
בוסטוס. וחב' הציעו שיטה אחת לרצף קטעים ארוכים של DNA שחוזר על עצמו.[42] השיטה משלבת שימוש בוקטור ליניארי לייצוב ו-exonuclease III למחיקה של אזורים עשירים ברצף פשוט מתמשך (SSRs). ראשית, קטעים עשירים ב-SSR משובטים לוקטור ליניארי שיכול לשלב ביציבות חזרות טנדם של עד 30kb. ביטוי של חזרות אסור על ידי מסימי התעתיק בווקטור. השלב השני כולל שימוש באקסונוקלאז III. האנזים יכול למחוק נוקלאוטיד בקצה 3' מה שגורם לייצור של מחיקה חד כיוונית של שברי SSR. לבסוף, מוצר זה אשר נמחק מקטעים מוכפל ומנתח עם מושבה PCR. הרצף נבנה לאחר מכן על ידי רצף מסודר של קבוצה של שיבוטים המכילים מחיקות שונות.
קישורים חיצוניים
הערות שוליים
- ↑ de Koning AP, Gu W, Castoe TA, Batzer MA, Pollock DD (בדצמבר 2011). "Repetitive elements may comprise over two-thirds of the human genome". PLOS Genetics. 7 (12): e1002384. doi:10.1371/journal.pgen.1002384. PMC 3228813. PMID 22144907.
{{cite journal}}: (עזרה) - ↑ "Repeated Sequence (DNA) - an overview | ScienceDirect Topics". www.sciencedirect.com. נבדק ב-2022-10-04.
- ^ 3.0 3.1 Lower SE, Dion-Côté AM, Clark AG, Barbash DA (בנובמבר 2019). "Special Issue: Repetitive DNA Sequences". Genes. 10 (11): 896. doi:10.3390/genes10110896. PMC 6895920. PMID 31698818.
{{cite journal}}: (עזרה) - ↑ McCLINTOCK B (1951-01-01). "Chromosome organization and genic expression". Cold Spring Harbor Symposia on Quantitative Biology. 16: 13–47. doi:10.1101/sqb.1951.016.01.004. PMID 14942727.
- ↑ Britten RJ, Kohne DE (באוגוסט 1968). "Repeated sequences in DNA. Hundreds of thousands of copies of DNA sequences have been incorporated into the genomes of higher organisms". Science. 161 (3841): 529–540. doi:10.1126/science.161.3841.529. PMID 4874239.
{{cite journal}}: (עזרה) - ↑ Shapiro JA, von Sternberg R (במאי 2005). "Why repetitive DNA is essential to genome function". Biological Reviews of the Cambridge Philosophical Society. 80 (2): 227–250. doi:10.1017/s1464793104006657. PMID 15921050.
{{cite journal}}: (עזרה) - ↑ Ohno S (1972). "So much "junk" DNA in our genome". Brookhaven Symposia in Biology. 23: 366–370. PMID 5065367.
- ↑ Orgel LE, Crick FH, Sapienza C (בדצמבר 1980). "Selfish DNA". Nature. 288 (5792): 645–646. Bibcode:1980Natur.288..645O. doi:10.1038/288645a0. PMID 7453798.
{{cite journal}}: (עזרה) - ↑ Palazzo AF, Gregory TR (במאי 2014). "The case for junk DNA". PLOS Genetics. 10 (5): e1004351. doi:10.1371/journal.pgen.1004351. PMC 4014423. PMID 24809441.
{{cite journal}}: (עזרה) - ↑ Joly-Lopez Z, Bureau TE (באפריל 2018). "Exaptation of transposable element coding sequences". Current Opinion in Genetics & Development. 49: 34–42. doi:10.1016/j.gde.2018.02.011. PMID 29525543.
{{cite journal}}: (עזרה) - ↑ "Tandem Repeat". Genome.gov (באנגלית). נבדק ב-2022-09-30.
- ↑ Sznajder ŁJ, Swanson MS (ביולי 2019). "Short Tandem Repeat Expansions and RNA-Mediated Pathogenesis in Myotonic Dystrophy". International Journal of Molecular Sciences. 20 (13): 3365. doi:10.3390/ijms20133365. PMC 6651174. PMID 31323950.
{{cite journal}}: (עזרה) - ↑ "MeSH Browser". meshb.nlm.nih.gov. נבדק ב-2022-09-30.
- ^ 14.0 14.1 Wahls WP (1998). "Meiotic recombination hotspots: shaping the genome and insights into hypervariable minisatellite DNA change". Current Topics in Developmental Biology. 37: 37–75. doi:10.1016/s0070-2153(08)60171-4. ISBN 9780121531379. PMC 3151733. PMID 9352183.
- ↑ Janssen A, Colmenares SU, Karpen GH (באוקטובר 2018). "Heterochromatin: Guardian of the Genome". Annual Review of Cell and Developmental Biology. 34 (1): 265–288. doi:10.1146/annurev-cellbio-100617-062653. PMID 30044650.
{{cite journal}}: (עזרה) - ^ 16.0 16.1 Qi J, Shafer RH (2005-06-02). "Covalent ligation studies on the human telomere quadruplex". Nucleic Acids Research. 33 (10): 3185–3192. doi:10.1093/nar/gki632. PMC 1142406. PMID 15933211.
- ↑ "Centromere". Genome.gov (באנגלית). נבדק ב-2022-09-30.
- ↑ Miga KH (בספטמבר 2015). "Completing the human genome: the progress and challenge of satellite DNA assembly". Chromosome Research. 23 (3): 421–426. doi:10.1007/s10577-015-9488-2. PMID 26363799.
{{cite journal}}: (עזרה) - ↑ Miga KH (בספטמבר 2015). "Completing the human genome: the progress and challenge of satellite DNA assembly". Chromosome Research. 23 (3): 421–426. doi:10.1007/s004390050508. PMID 26363799.
{{cite journal}}: (עזרה) - ↑ Padeken J, Zeller P, Gasser SM (באפריל 2015). "Repeat DNA in genome organization and stability". Current Opinion in Genetics & Development. Genome architecture and expression. 31: 12–19. doi:10.1016/j.gde.2015.03.009. PMID 25917896.
{{cite journal}}: (עזרה) - ↑ "Interspersed repetitive sequences - Latest research and news | Nature". www.nature.com. נבדק ב-2022-09-30.
- ^ 22.0 22.1 22.2 22.3 Wicker T, Sabot F, Hua-Van A, Bennetzen JL, Capy P, Chalhoub B, et al. (בדצמבר 2007). "A unified classification system for eukaryotic transposable elements". Nature Reviews. Genetics. 8 (12): 973–982. doi:10.1038/nrg2165. PMID 17984973.
{{cite journal}}: (עזרה) - ^ 23.0 23.1 23.2 23.3 Nicolau M, Picault N, Moissiard G (באוקטובר 2021). "The Evolutionary Volte-Face of Transposable Elements: From Harmful Jumping Genes to Major Drivers of Genetic Innovation". Cells. 10 (11): 2952. doi:10.3390/cells10112952. PMC 8616336. PMID 34831175.
{{cite journal}}: (עזרה) - ^ 24.0 24.1 Kramerov DA, Vassetzky NS (2011). "SINEs". Wiley Interdisciplinary Reviews. RNA. 2 (6): 772–786. doi:10.1002/wrna.91. PMID 21976282.
- ↑ Lee HE, Ayarpadikannan S, Kim HS (2015). "Role of transposable elements in genomic rearrangement, evolution, gene regulation and epigenetics in primates". Genes & Genetic Systems. 90 (5): 245–257. doi:10.1266/ggs.15-00016. PMID 26781081.
- ↑ Mangiavacchi A, Liu P, Della Valle F, Orlando V (ביולי 2021). "New insights into the functional role of retrotransposon dynamics in mammalian somatic cells". Cellular and Molecular Life Sciences. 78 (13): 5245–5256. doi:10.1007/s00018-021-03851-5. PMC 8257530. PMID 33990851.
{{cite journal}}: (עזרה) - ↑ Ichiyanagi K (2013). "Epigenetic regulation of transcription and possible functions of mammalian short interspersed elements, SINEs". Genes & Genetic Systems. 88 (1): 19–29. doi:10.1266/ggs.88.19. PMID 23676707.
- ↑ Pearson CE, Zorbas H, Price GB, Zannis-Hadjopoulos M (באוקטובר 1996). "Inverted repeats, stem-loops, and cruciforms: significance for initiation of DNA replication". Journal of Cellular Biochemistry. 63 (1): 1–22. doi:10.1002/(SICI)1097-4644(199610)63:1<1::AID-JCB1>3.0.CO;2-3. eISSN 1097-4644. PMID 8891900.
{{cite journal}}: (עזרה) - ^ 29.0 29.1 Usdin K, House NC, Freudenreich CH (22 בינואר 2015). "Repeat instability during DNA repair: Insights from model systems". Critical Reviews in Biochemistry and Molecular Biology. 50 (2): 142–167. doi:10.3109/10409238.2014.999192. PMC 4454471. PMID 25608779.
{{cite journal}}: (עזרה) - ^ 30.0 30.1 Massey TH, Jones L (בינואר 2018). "The central role of DNA damage and repair in CAG repeat diseases". Disease Models & Mechanisms. 11 (1): dmm031930. doi:10.1242/dmm.031930. PMC 5818082. PMID 29419417.
{{cite journal}}: (עזרה) - ↑ Cattaneo E, Zuccato C, Tartari M (בדצמבר 2005). "Normal huntingtin function: an alternative approach to Huntington's disease". Nature Reviews. Neuroscience. 6 (12): 919–930. doi:10.1038/nrn1806. PMID 16288298.
{{cite journal}}: (עזרה) - ↑ Maiuri T, Mocle AJ, Hung CL, Xia J, van Roon-Mom WM, Truant R (בינואר 2017). "Huntingtin is a scaffolding protein in the ATM oxidative DNA damage response complex". Human Molecular Genetics. 26 (2): 395–406. doi:10.1093/hmg/ddw395. PMID 28017939.
{{cite journal}}: (עזרה) - ↑ Schulte J, Littleton JT (בינואר 2011). "The biological function of the Huntingtin protein and its relevance to Huntington's Disease pathology". Current Trends in Neurology. 5: 65–78. PMC 3237673. PMID 22180703.
{{cite journal}}: (עזרה) - ↑ Penagarikano O, Mulle JG, Warren ST (2007-09-01). "The pathophysiology of fragile x syndrome". Annual Review of Genomics and Human Genetics. 8 (1): 109–129. doi:10.1146/annurev.genom.8.080706.092249. PMID 17477822.
- ↑ Hagerman RJ, Berry-Kravis E, Hazlett HC, Bailey DB, Moine H, Kooy RF, et al. (בספטמבר 2017). "Fragile X syndrome". Nature Reviews. Disease Primers. 3 (1): 17065. doi:10.1038/nrdp.2017.65. PMID 28960184.
{{cite journal}}: (עזרה) - ^ 36.0 36.1 Abugable AA, Morris JL, Palminha NM, Zaksauskaite R, Ray S, El-Khamisy SF (בספטמבר 2019). "DNA repair and neurological disease: From molecular understanding to the development of diagnostics and model organisms". DNA Repair. 81: 102669. doi:10.1016/j.dnarep.2019.102669. PMID 31331820.
{{cite journal}}: (עזרה) - ↑ Honti V, Vécsei L (ביוני 2005). "Genetic and molecular aspects of spinocerebellar ataxias". Neuropsychiatric Disease and Treatment. 1 (2): 125–133. doi:10.2147/nedt.1.2.125.61044. PMC 2413192. PMID 18568057.
{{cite journal}}: (עזרה) - ↑ Bürk K (2017). "Friedreich Ataxia: current status and future prospects". Cerebellum & Ataxias. 4 (1): 4. doi:10.1186/s40673-017-0062-x. PMC 5383992. PMID 28405347.
- ↑ Mazzara PG, Muggeo S, Luoni M, Massimino L, Zaghi M, Valverde PT, et al. (באוגוסט 2020). "Frataxin gene editing rescues Friedreich's ataxia pathology in dorsal root ganglia organoid-derived sensory neurons". Nature Communications. 11 (1): 4178. Bibcode:2020NatCo..11.4178M. doi:10.1038/s41467-020-17954-3. PMC 7442818. PMID 32826895.
{{cite journal}}: (עזרה) - ↑ Hahn C, Salajegheh MK (בינואר 2016). "Myotonic disorders: A review article". Iranian Journal of Neurology. 15 (1): 46–53. PMC 4852070. PMID 27141276.
{{cite journal}}: (עזרה) - ↑ van Blitterswijk M, DeJesus-Hernandez M, Rademakers R (בדצמבר 2012). "How do C9ORF72 repeat expansions cause amyotrophic lateral sclerosis and frontotemporal dementia: can we learn from other noncoding repeat expansion disorders?". Current Opinion in Neurology. 25 (6): 689–700. doi:10.1097/WCO.0b013e32835a3efb. PMC 3923493. PMID 23160421.
{{cite journal}}: (עזרה) - ^ 42.0 42.1 De Bustos A, Cuadrado A, Jouve N (בנובמבר 2016). "Sequencing of long stretches of repetitive DNA". Scientific Reports. 6 (1): 36665. Bibcode:2016NatSR...636665D. doi:10.1038/srep36665. PMC 5098217. PMID 27819354.
{{cite journal}}: (עזרה) - ↑ Slotkin RK (1 במאי 2018). "The case for not masking away repetitive DNA". Mobile DNA. 9 (1): 15. doi:10.1186/s13100-018-0120-9. PMC 5930866. PMID 29743957.
{{cite journal}}: (עזרה)
רצף חוזר35458045Q424473


